iDES促进 CAPP-Seq提高ctDNA灵敏性,促进其在临床中的应用
ctDNA具有无创性、可重复性监测肿瘤的优点,且大量一期和二期临床研究数据已证明ctDNA在临床肿瘤的早期诊断、疗效观察、预后评估及转移风险分析等具有巨大的应用价值。但由于cfDNA含量少,检测背景误差大,造成检测的灵敏性和特异性低,限制了ctDNA检测在临床的应用。
2014年研究人员在Nature Medicine上发表了一篇文章介绍了一种全新的ctDNA检测技术,CAPP-Seq(肿瘤个体化分析深度测序法),作者在肿瘤基因突变数据库(COSMIC)寻找复发突变相关的外显子,再从肿瘤基因图谱库的407位NSCLC患者全基因组测序结果筛选,设计的突变点覆盖超过了95%的NSCLC。后应用一种大化迭代算法设计出NSCLC ctDNA的“筛选器“。对样品进行靶向捕获后进行深度测序。本方法灵敏度高,特异性强且经济可行的优点。
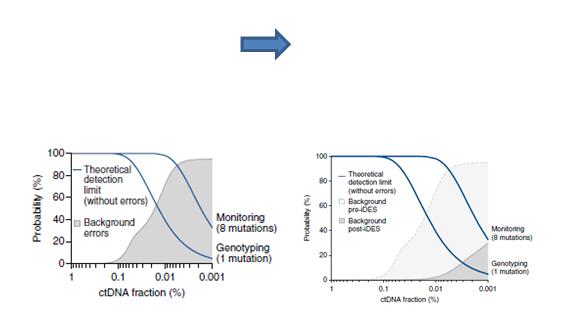
通过CAPP-Seq检测ctDNA等位基因频率在0.2%本底误差开始逐渐明显,而达到CAPP-Seq检测极限0.02%时人工误差达到50%,因此降低背景误差尤为重要。因此作者对本方法进行了改善,结果于2016年发表在Nature biotechnology上,作者引进了iDES(集成数字误差抑制)的方法,它是将条形码技术和背景抛光结合起来的一种方法,它们能分别使CAPP-seq敏感性提升3倍,而结合后提升15倍,通过iDES改善后CAPP-Seq能够检测到频率低至0.004%的突变等位基因。
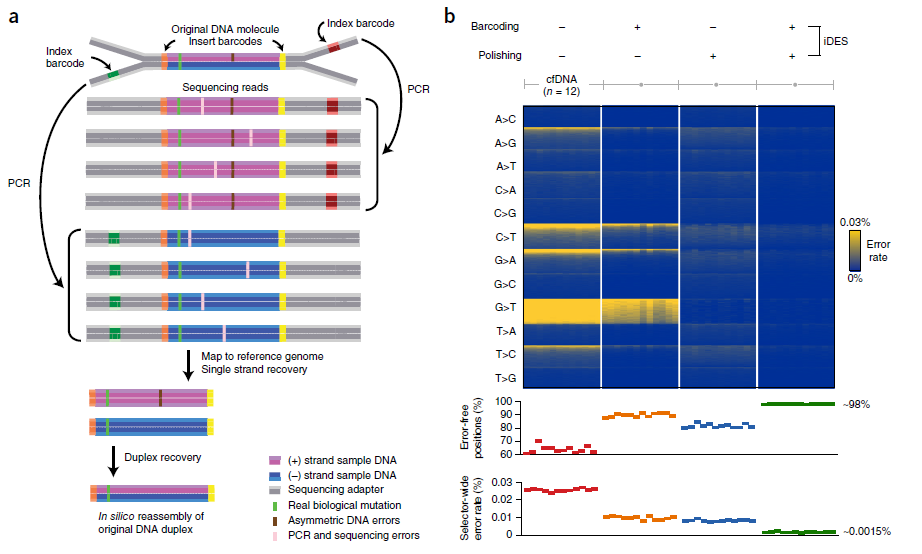
条形码技术是利用两个插入条码、两个索引条码及适配器组装双链DNA,分别扩增,后得到相对较纯反义链,再重新组装成拥有真实突变的双链DNA。结合条码技术及背景抛光协同使ctDNA检测的无错误位点从90%升至98%,全误差率低至0.0015%。
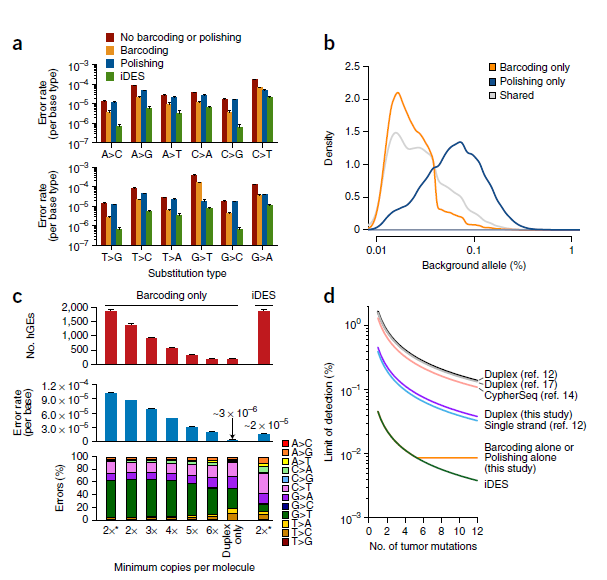
背景抛光可以修正背景误差,条形码技术可以修正随机误差,这两个技术结合起到一个互补的作用,使iDES错检率较低。iDES在低拷贝数下就可以达到较低的错检率。通过与其他文献报答方法的比较可以看出,iDES检出限明显低于其他方法。
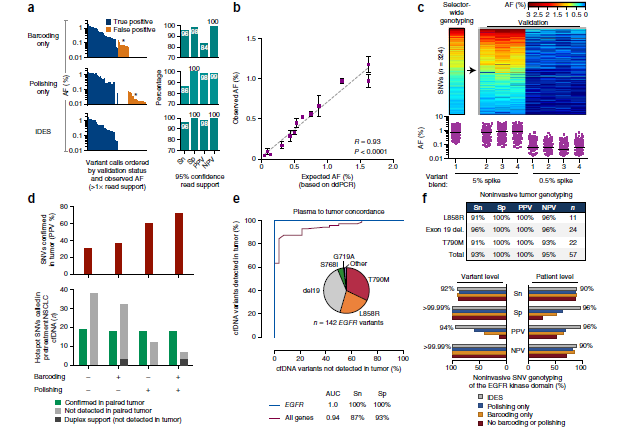
作者对iDES敏感性、特异性、阳性预测值、阴性预测值检测,iDES表现都是好的。通过与ddPCR对比,发现iDES与ddPCR等位基因频率有较高的一致性。检测肿瘤热点突变中iDES能使阳性预测值PPV提升至70%,并且能使肿瘤配对样本中没有检测的cfDNA达到低。在检测EGFR突变中发现敏感性和特异性同时达到100%,而针对不同突变L858R、Exon19 del、T790M时特异性和PPV达到100%,敏感性也有91%~96%。
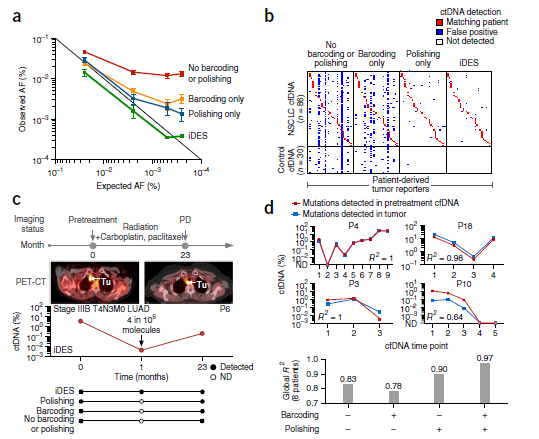
在临床实验上iDES也表现出较好的效果,作者通过将突变的胶质瘤与正常细胞cfDNA混合的,观测iDES的检测效果,iDES表现出较高的一致性。iDES能监测NSCLC患者105cfDNA中4个ctDNA。这是其他方法无法检出的。作者还利用肺癌患者血浆中与肿瘤组织检测到的突变基因进行了回归分析,iDES的相关性可以到达0.97。
总的来说iDES可以使CAPP-seq可检测100%的II~IV期NSCLC,I期检测是50%,该方法特异性高达96%,能检测突变等位基因频率低至0.004%,ctDNA水平与肿瘤大小高度一致性,能反应残余病灶及治疗相关的影像学反应,并且比影像学更加灵敏,iDES能是从血中检测EGFR激酶区域突变谱敏感性达到92%,特异性达到99.99%,在患者水平其敏感性和特异性分别是90%和96%,iDES能监测NSCLC患者105cfDNA中4个ctDNA。
我们可以预测iDES促进CAPPSeq将辅助非侵入性基因分型及在临床中发挥其优势。
